![]()
分子モデリング、ドラッグデザイン、インシリコ創薬支援システム:タンパク質ホモロジーモデリングソフト
![]()

国産初のタンパク質ホモロジーモデリングソフト:複雑な生体高分子システムの立体構造精密化のための全機能搭載、生体高分子の量子化学計算もワンクリックで実施、鋳型がなくてもAlpha Foldと組み合わせて高精度モデルが簡単に作成可能
Homology Modeling Professional for HyperChem, Revision H1
with Gaussian Interface for HyperChem & ONIOM Interface for Receptor
Compatible to NAMD Molecular Dynamics and ABINIT-MP / GAMESS Fragment Molecular Orbital Quantum Mechanics Calculations for Entire Molecular System

![]()
![]()
Gaussian, GaussView, NAMD, VMD 互換機能搭載、ABINIT-MP, GAMESS ファイル互換機能搭載
HMHC:新型コロナウイルスに対する世界初大規模仮想スクリーニング成果を達成、国内最初の抗SARS-CoV-2薬医薬品候補リスト論文発表を達成(2020年5月1日)
HMHCを用いた受託研究サービスがご利用いただけます。多数の実績がありますので、是非導入前にご検討ください。
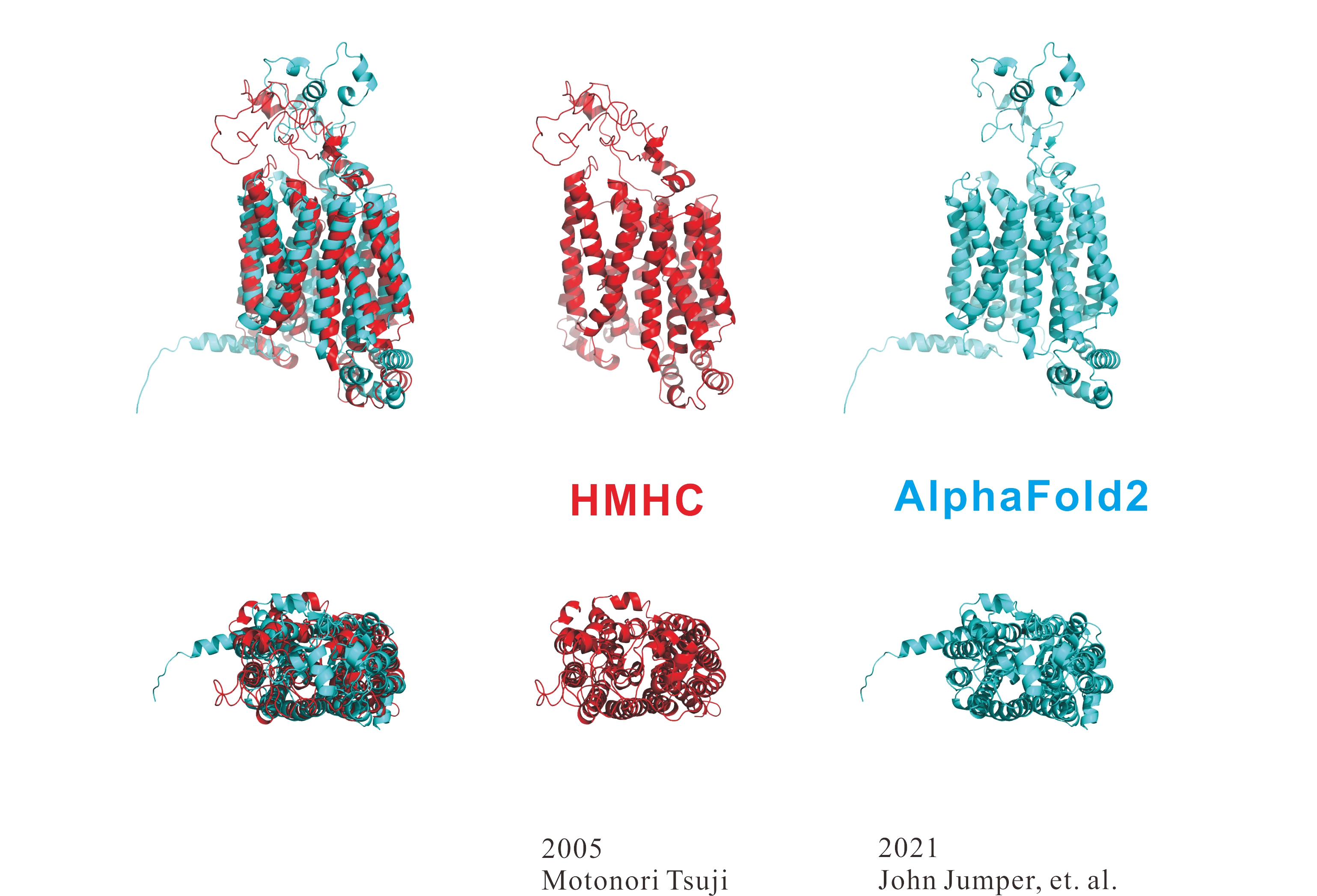
AlphaFold2 vs Homology Modeling Professional for HyperChem(HMHC)
下図は、アミノ酸配列が報告されてから20年以上もファミリーも含めて立体構造が不明なタンパク質についてHomology Modeling Professional for HyperChem(HMHC)とAlphaFold2で立体構造を予測した結果です。
HMHC(2005年公開)はAlphaFold2(2021年公開)より16年も前に公開していますが、膜貫通部分のアラインメントは一致しており、HMHCとAlphaFold2から得られたホモロジーモデルは熱力学的に複数のコンフォメーションを採用すると予測される膜外ループ構造が異なる程度でした。
本蛋白質は立体構造未知のため、どちらのホモロジーモデルが正解に近いかは現時点では不明ですが、HMHCはAlphaFold2と同様のモデルを提供できます。HMHCは側鎖ロータマーや生体高分子に含まれる結晶水や低分子までも高精度にモデリングできる点でアドバンテージがあります。加えて、HMHCではヘテロダイマーなどの高次構造や核酸との複合体モデルなども簡単にモデリングでき、量子化学計算プログラムGaussian(ONIOM計算)や分子動力学シミュレーションプログラムNAMDともシームレスに連携できます。AlphaFold2で主鎖構造を予測しておき、HMHCで側鎖立体配座や全体構造を精密化するといった方法論も有効と考えられます。
最新情報 最新バージョンは8.1.5(2024/7/1) 追加機能と今後のロードマップを公開
Homology Modeling Professional for HyperChem, Revision H1がインシリコ創薬統合プラットフォームとして更に進化。
量子力学計算Gaussian ONIOM Interface大幅機能強化、分子動力学計算NAMD(VMD) CHARMM-basedファイル互換機能追加、フラグメント分子軌道計算ABINIT-MP(BioStation Viewer)/ GAMESS(Fu/Facio)ファイル互換機能追加
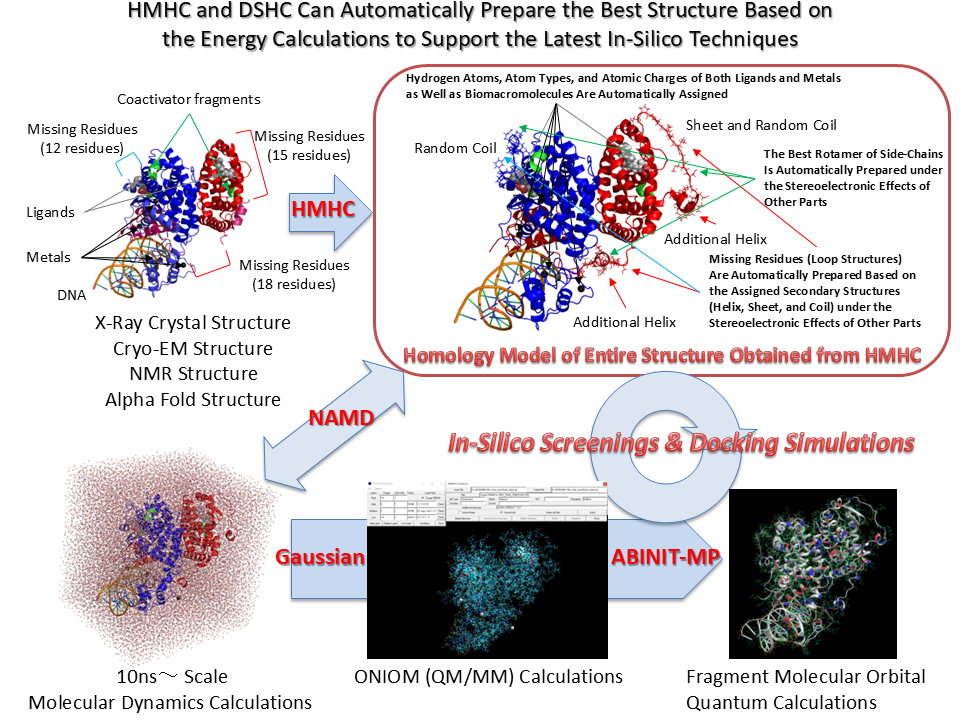
最先端のインシリコ創薬において、生体高分子システムなどの巨大分子システムに対して全系量子力学計算を実施する場合、分子力学計算による構造最適化計算や分子動力学計算で用意した初期構造では一点計算さえ収束しないか、異常なエネルギー値を与えます。フラグメント分子軌道法などで全系量子力学計算を実施する場合は、ONIOM法で予め初期構造を更に構造最適化して置く必要があります。ONIOM Interface for ReceptorはHomology Modeling Professional for HyperChemやDocking Study with HyperChemで準備した高精度初期構造(複合体構造)を量子力学計算用に更に精密化して全系量子力学計算を成功させる最善のソリューションを提供します。
Motonori Tsuji*, Koichi Shudo, Hiroyuki Kagechika. FEBS Open Bio., 7, 391-396, 2017, DOI: 10.1002/2211-5463.12188.
最強の論理的分子設計ツールHyperChemとGaussianで生体高分子モデリング、解析、シミュレーション
様々な状態の生体高分子システム(タンパクおよび核酸分子システム)に対応
HyperChem、Gaussian計算エンジンによるエネルギーベースの最先端手法
高精度タンパク質二次構造予測アルゴリズムによる第一原理での立体構造予測機能搭載
ホモロジーモデリング過程での、挿入配列モデリング、主鎖モデリング、側鎖モデリング、共有結合低分子モデリング、ヘテロ分子・金属原子影響下モデリング、ホモ・ヘテロ会合状態モデリング、溶媒和条件下モデリングなど従来製品では利用できない最先端手法がエネルギーベースで実施できます。
なお、タンパクホモロジーモデリング作業だけの場合は主鎖、側鎖、全体構造の最適化に関する3つのモジュールプログラムを利用するだけです。また、これらモジュール内の作業は自動化されているため、ほぼ全自動で高精度にホモロジーモデリングできます。
Powered by Homology Modeling Professional for HyperChem THE IMPOSSIBLE BECOMES POSSIBLE.
最新、高性能・高機能生体高分子システム分子モデリング、機能解析、シミュレーションパッケージ
分子力学計算はもとよりab initio量子化学計算、分子動力学シミュレーションを網羅的に実施して、巨大分子モデリング、機能解析、シミュレーションできる、HyperChemをコアとする最先端分子モデリングパッケージ。HyperChemとGaussianをシームレスに制御、計算結果をリアルタイムにフィードバック。完全自動ONIOMインターフェイスにより巨大分子システム全体を量子化学的に処理。すべての操作が完全自動化・GUI化され、構造ベース創薬からライフサイエンス研究における論理的分子設計、ドラッグデザイン、未踏研究をロジカルに支援。さらに、世界初、究極の革新技術を搭載したin silicoスクリーニング対応生体高分子-リガンドフレキシブルドッキングシミュレーションプログラム、Docking Study with HyperChemパッケージと高度に連携。リビジョンB1からは完全自動レンダリング機能、ラベリング機能を全モジュールに搭載して、文献のフィギュアレベルの美しさで分子モデリング、機能解析、シミュレーションが可能。
導入実績
国内企業(医薬品、食料品、化学、科学計算、電気機器、輸送用機器)、国内国立研究機関、国内大学研究機関(医学部、薬学部、農学部、理学部、工学部)、海外企業、海外大学研究機関
最新パンフレット(PDFファイル:1MB) 旧パンフレットと併せてご利用ください
リビジョンG1パンフレット(PDFファイル:4MB) 旧パンフレットと併せてご利用ください
パンフレット(PDFファイル:12MB) 旧パンフレットです
SBDDソリューション資料(PDFファイル:2MB)
製品紹介論文
Motonori Tsuji*. Molecular Science, 1, NP004, 2007.
成果学会報告
成果論文
Microbiol. Spectrum. 12, 2024.
J. Autoimmun. 139, 103085, 2023.
Motonori Tsuji*. Int. J. Mol. Sci. 23, 11009, 2022.
PLOS ONE 16, e0257705, 2021.
Chem. Pharm. Bull. 68, 1193-1200, 2020.
J. Virol. 2020.
Motonori Tsuji*. FEBS Open Bio., 10, 995-1004, 2020.
J. Pharmacol. Exp. Ther. 372, 277-284, 2020.
Motonori Tsuji*. J. Comput. Aided Mol. Des., 31, 577-585, 2017, DOI: 10.1007/s10822-017-0025-6.
Motonori Tsuji*, Koichi Shudo, Hiroyuki Kagechika. FEBS Open Bio., 7, 391-396, 2017, DOI: 10.1002/2211-5463.12188.
Motonori Tsuji*. J. Mol. Graph. Model., 62, 262-275, 2015.
Motonori Tsuji*, Koichi Shudo, Hiroyuki Kagechika. J. Comput. Aided Mol. Des., 29, 975-988, 2015.
Motonori Tsuji*. J. Struct. Biol, 185, 355-365, 2014.
Chem-Bio Informatics Journal, 12, 1-13, 2012.
Plant Cell Physiol. 2012 Jul; 53(9): 1638-1647.
Biochemistry. 2010 Dec; 49(50): 10647-10655.
Biochemical and Biophysical Research Communications. 2010 Apr 30; 395(2): 173-177.
Science. 2008 Feb 1; 319(5863): 624-7.
世界が認める最強の分子モデリング環境
HyperChem front-ended
世界が認める最強の計算化学環境
Gaussian back-ended
本製品はGaussianオフィシャルサイトにトップレベルでリンクされています。
Homology Modeling Professional for HyperChem概要
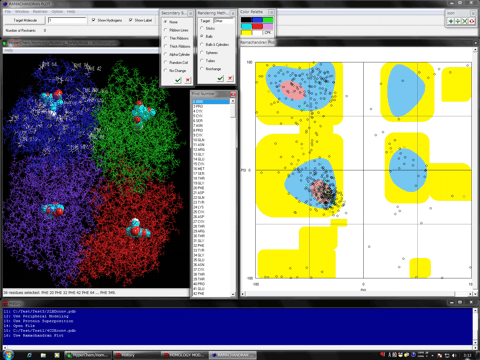
Homology Modeling Professional for HyperChemは、分子モデリング・分子シミュレーションソフトで世界的に定評のあるHyperChemをフロントエンドとし、生体高分子モデリング、機能解析およびシミュレーションに必要な多くのモジュールプログラムから構成されています。これらモジュールプログラムの多くが既知モデリングシステムにはない最先端独自技術を搭載しており、HyperChemの機能と融合することで、かつてない画期的な分子モデリング、機能解析、シミュレーション支援システムおよび新薬開発研究における創薬支援システムとなります。
研究者による研究者のためのソフトウエア設計
Homology Modeling Professional for HyperChemでは直感的操作性に強く配慮されており、HyperChemオプションでは複雑なステップを必要とする操作がワンクリックで達成されます。ほとんどの場合、ボタンを選択するだけで自動的に目的が達成されるように設計されています。また、Homology Modeling Professional for HyperChemで用意されるツールにより、モデリング中のHyperChem自体の操作は分子の回転操作以外を必要としません。完全自動レンダリング機能および自動ラベルリング機能を全モジュールプログラムに搭載しており、文献のフィギュアやデモンストレーションレベルの美しさでモデリング、機能解析、シミュレーションできます。その他、分子番号参照時における生体高分子(タンパク質分子および核酸分子)と他の分子との区別、目的部位へのセンタリング機能、モデリングに伴う構造修正時の取り消し機能、誤った操作に対するUndo機能などの細かなサポートがなされています。このため、HyperChemのコマンドおよびオプションに関する予備知識なしでも生体高分子モデリングが可能であるとともに、パワーユーザーはHyperChemの機能もあわせて駆使できるよう設計されています。
複雑な分子システムに対応
Homology Modeling Professional for HyperChemでは、扱える原子数が約10万(HyperChem7.5以降)と、現在PDBに登録されている構造データを網羅的に扱えます。さらに、生体高分子以外の高分子、低分子あるいは金属原子等を含む混在環境のまま、生体高分子モデリング、機能解析、シミュレーションできます。また、これらが生体高分子と共有あるいは配位結合している場合にも対応しています。必要なパラメータは自動的に割り振られるか、後述する各種計算化学により生成できます。このため、本システムで作成した高精度生体高分子モデルまたは精密化した結晶構造とシードやリード化合物との詳細な相互作用解析により、化合物最適化など高度なin silico創薬研究が可能となります。
世界最強計算化学環境:分子力学計算から量子化学計算まで
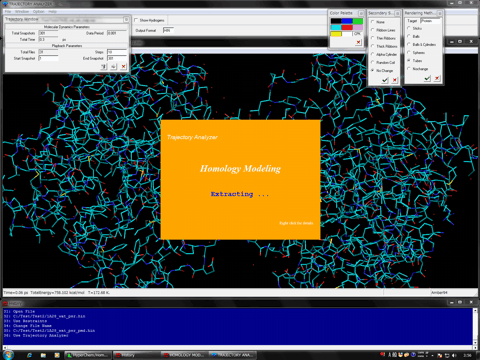
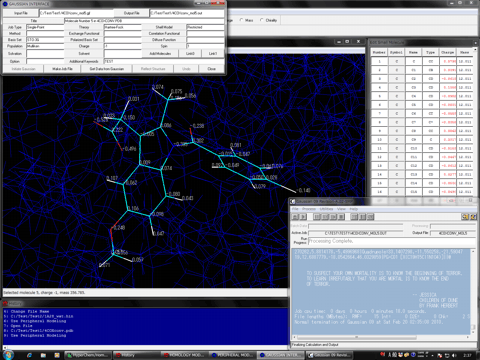
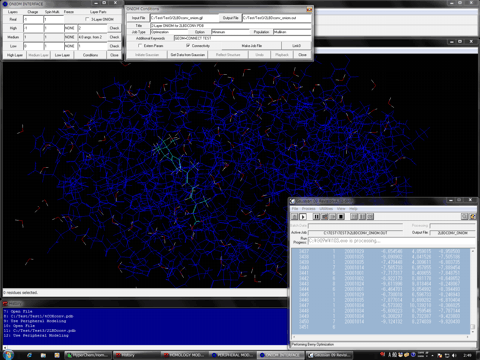
HyperChemでサーポートされる計算化学がすべて利用できることから、極めて強力な生体高分子機能解析環境を提供します。Mopac2000とMopac Interface for HyperChemおよびQ-Chem1.2 for Windowsが利用可能な環境ではこれらがサポートする計算化学がすべて利用できます。さらに、Homology Modeling Professional for HyperChemに搭載されるGaussian Interface for HyperChemおよびGaussianが搭載するONIOM計算のための完全自動インターフェイスであるONIOM Interface for Receptorを介してGaussianジョブを実行し、生体高分子結晶構造中に含まれるすべての分子種の電子状態を自動的、リアルタイムにHyperChem上の分子システムに反映させることができ、これにより分子システム全体を量子化学的に処理して、究極のモデリング、機能・相互作用解析、シミュレーションさらには未踏研究をもロジカルに支援します。最新版では分子動力学計算プログラムNAMD(VMD)、全系量子化学計算用フラグメント分子軌道計算プログラムABINIT-MP(BioStation Viewer)およびGAMESS(Fu/Facio)と連携するための互換機能を搭載しています。
即研究可能
PDBファイル、パラメータファイル等を一切編集、準備する必要がないため、研究者は本来の研究に即打ち込めます。ヘビーユーザーは自動生成される入力ファイルのコマンドをテキストベースで詳細にチューニングすることもできます。プログラムが使用したパラメータおよび結果を含む詳細なログが自動的に生成されます。モデリングおよび機能解析に使用したファイルの管理をプログラム側が行います。HyperChemバージョン5.x/6.x/7.x/8.0xをサポートしていることから、初期バージョンのHyperChemが最新の生体高分子モデリング機能解析環境として利用できるようになります。
画期的パフォーマンス
Homology Modeling Professional for HyperChemは単なる創薬支援ツールとしてのみならず、同種既存システムでは不可能な、レセプタータンパクによるリガンド認識メカニズムなどの未踏の研究をもサポートできる優れた潜在的パフォーマンスを有しており、かつ論理的な研究の場を提供します。IntelベースWindowsプラットフォームで動作するため、計算速度、ユーザーインターフェイスの両面でもUnixをプラットフォームとするシステムとは圧倒的性能差が期待できます。
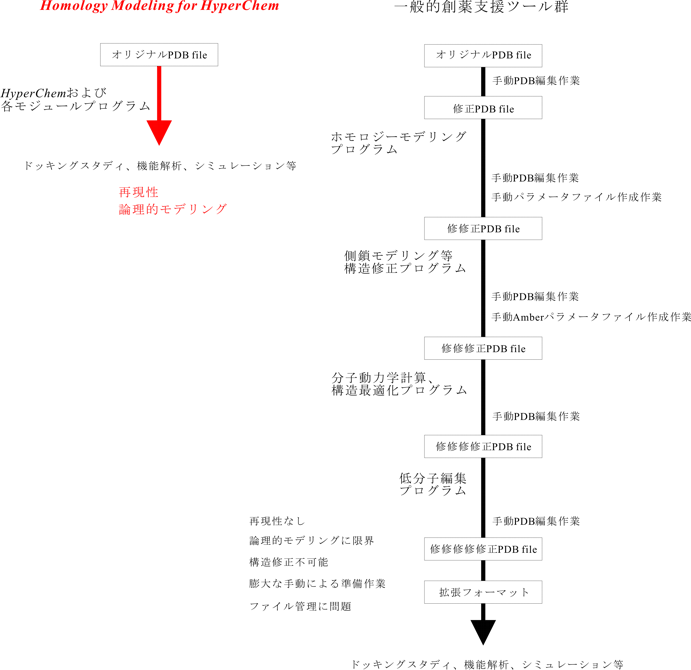
再現性と論理的モデリング
Homology Modeling Professional for HyperChemは分子モデリングを一貫してHyperChem上で行うことにより、サイエンスの基本である再現性と論理性を確保するとともに、作業時間の大幅な短縮を可能にしています。
Homology Modeling Professional for HyperChemはHyperChemをフロントエンドとし、低分子設計の代表的論理的ツールであるHyperChemとGaussianの機能を余すことなく利用してタンパク質分子システムのホモロジーモデリングと構造精密化を論理的に実施できる究極の分子設計支援システムです。Homology Modeling Professional for HyperChemでは、他の同種既存システムがいまなお知識ベースで実施している作業をすべてエネルギー計算に基づいて論理的に実施できるだけでなく、結果として網羅性、効率性、再現性までもが保証されます。具体的には、Homology Modeling Professional for HyperChemパッケージにおける個々のモジュールプログラムはモデルに最善の初期構造を与えるために利用します。これは、計算化学では計算理論レベルに関係なく、計算対象である初期構造の良し悪しによってのみ、信頼できる、つまりグローバルミニマムの最終構造を与えるか否かが決まるからです。各モジュールプログラムで論理的にモデリングされた最善の初期構造をもって最終的に分子動力学アニーリングおよび構造最適化計算を実施して最良の最終構造を調整します。こうした作業には非常に高度な専門知識と技術が要求されますが、Homology Modeling Professional for HyperChemではこれらの作業がほぼ自動化されており、ユーザーは用意されているボタンをクリックするだけで目的を達成できるように設計されています。その上でさらに、個々の研究者のロジックで本質的な作業のみに集中して分子設計するための環境までもが提供されています。
具体的なHomology Modeling Professional for HyperChemパッケージの使用例に関しては、ホモロジーモデリングチュートリアルをご参照ください。
最新のテスト情報およびリリースノートは技術サポートにあります。
モジュールプログラム
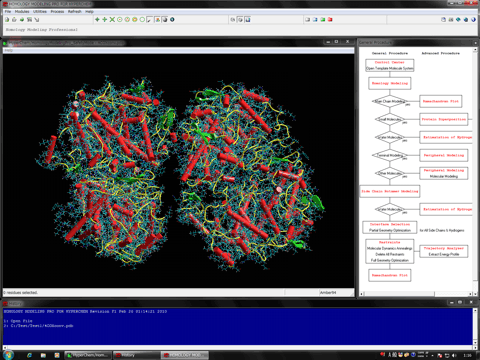
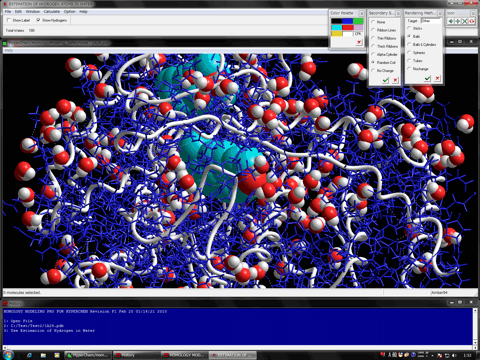
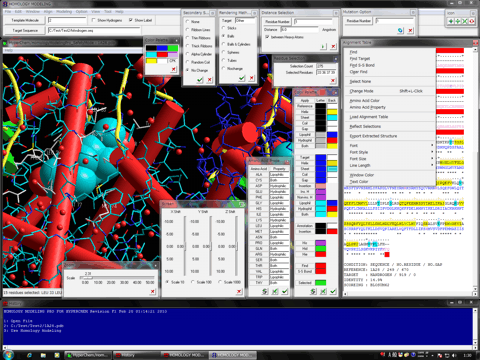
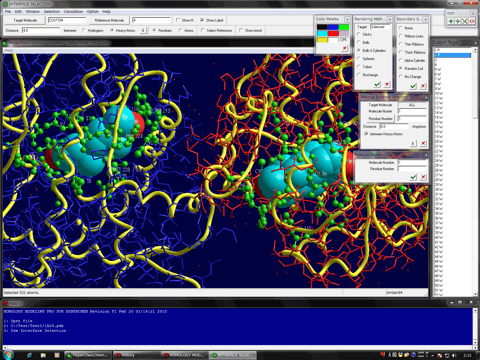
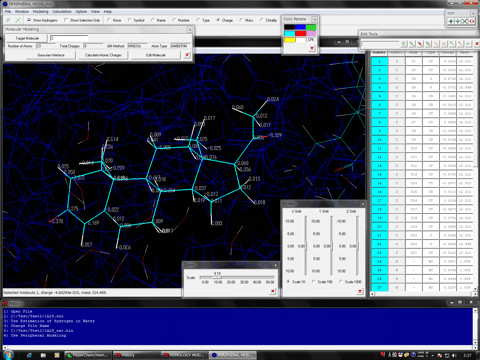
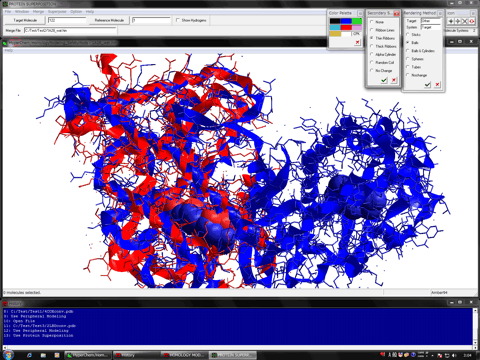
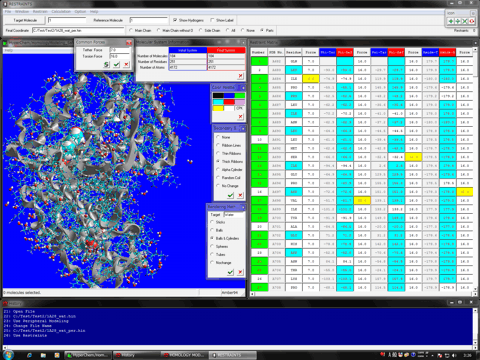
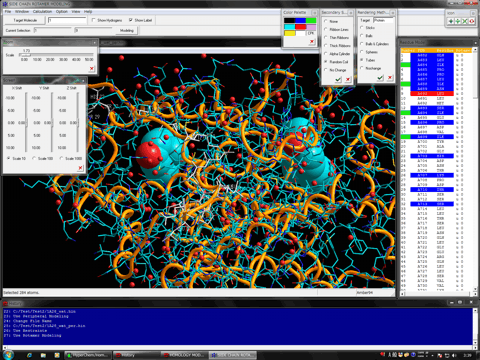
Gaussian Interface for HyperChem
推奨最小システム構成
プロセッサー: マルチコアIntel CoreまたはXeon 2GHz以上(Intel Pentium III以降)
オペレーティングシステム: Microsoft Windows 10及び11(Microsoft Windows 95以降、全機能を利用するにはXP以降が必要)
メモリー (RAM): 8 GB (OINOM計算を実施する場合は64GB)以上(最低256 MB以上)
モニタ:SXGA以上(フルHD以上を推奨)
グラフィックスボード:OpenGL対応ボードを推奨
マウス:キーストロークを割り当てられる多機能マウスを推奨
その他: HD 512GB(OINOM計算を実施する場合は2TB)以上を推奨; CD-ROMドライブ、ネットワークカード
必要なソフトウェア
必須
HyperChem(Windows版;Student Versionは除く):HyperChem Release 8.0.10 Professional for Windows (Release 5.1.1以降、全機能を利用するにはRelease 7.5.2以降が必要)
TclPro1.2(Windows版):Homology Modeling Professional for HyperChemの使用にはTclPro1.2(Windows版)が必要です。TclPro1.2はウェブ上から無料で入手できます。
オプション
Gaussian(Windows版またはUnix版): Gaussian Interface for HyperChemおよびONIOM Interface for Receptorを使用してその場でジョブ結果を反映する場合はGaussian16 64ビット マルチプロセッサ版(Gaussian98 リビジョンA9以降 64ビットまたは32ビット、マルチプロセッサ版またはシングルプロセッサ版が必要)
OpenBabel(Windows版、64ビット版または32ビット版):HIN形式(HyperChemフォーマット)またはPDB形式からMDL SDF形式、Tripos MOL2あるいはAutoDock PDBQT形式で出力する場合に必要
ホモロジー検索機能を利用する場合はWindows版ClustalW、BlastおよびFastaが必要
連携
その他、ナノ秒スケールの分子動力学計算を実施する場合はNAMDおよびVMD、全系量子力学計算を実施する場合はABINIT-MPおよびBioStation Viewer、GAMESSおよびFuまたはFacioを各ソフトウエアのライセンスに基づいて各自の責任において用意してください。
備考
PDBフォーマットバージョン2.3、3.0-3.3対応
Homology Modeling Professional for HyperChemはTcl/Tkプログラムと32ビットWindowsアプリケーションからなるソフトウェアパッケージです。
このソフトウェアパッケージはTclPro1.2コンパイラーでコンパイルされています。そのため、ソフトウェアパッケージの使用には、パッケージのインストールに先立って、TclPro1.2のインストールが必要です。
充実したオンラインヘルプがソフトウェアパッケージの個々のプログラムから使用できます(英文)。
全機能を実践チュートリアル形式で紹介した日本語版ユーザーマニュアル1巻と2巻(同種既存システムのマニュアルはもちろん、文献、書籍からも絶対に得られない最先端ノウハウを満載し、文章だけで133ページおよび34ページにわたる詳細解説)が付属します。
コントロールセンターを使ったホモロジー検索作業から基本的なホモロジーモデリング作業を初心者用にフィギュア付きで紹介した日本語リファレンスマニュアル(22ページ)が付属します。
重要:ソフトウェアパッケージをインストールするにあたり、まずシステムにHyperChemとTclPro1.2がインストールされていることを確認してください。このパッケージにはHyperChemおよびGaussianは含まれていません。
*HyperChemはHypercube, Incの登録商標です。
**GaussianはGaussian, Inc.の登録商標です。
***本製品の開発にあたり、Hypercube, Inc.およびGaussian, Inc.の承認済みです。













