![]()
新製品
What's New in Revision H1. 最新バージョンは8.1.5(2024/7/1)です。
インシリコ創薬統合プラットフォームへと進化
Docking Study with HyperChem
Essential, Premium Essential, Professional, Advanced, Ultimate, and Cluster
with AutoDock Vina In Silico Screenings Interface
Compatible to rDock and GNINA Docking Simulations, NAMD Molecular Dynamics, and ABINIT-MP / GAMESS Fragment Molecular Orbital Quantum Mechanics Calculations for Entire Molecular System
旧リビジョンからの変更点
AutoDock Vinaインシリコスクリーニングに必要な全作業、化合物データベース整備−各種ファイル準備−実行−結果閲覧−ヒット絞り込み、に関する機能を各モジュールプログラムに搭載、パッケージグレードに関係なく化合物数無制限のインシリコスクリーニングをサポート、AutoDockおよびAutoDock Vina単独で実施したドッキングモードの閲覧にも利用可能
rDockやGNINAなどSDF形式でドッキング結果を出力するドッキングプログラムからのドッキング結果の解析、結果閲覧、ヒット絞り込み機能搭載(rDockやGNINAのドッキングモードを初期構造として引き続き、AutoDockまたはAutoDock Vinaでドッキングが実施できる)
フラグメント分子軌道計算プログラムABINIT-MP(BioStation Viewer)およびGAMESS(Fu/Facio)用PDB形式への変換機能を搭載、全系量子力学計算プログラムとシームレスに連携可能
分子動力学計算プログラムNAMDのトラジェクトリからVMDで抽出したCHARMMベースのPDB形式を通常のPDB形式に変換する機能を搭載、数十ナノ秒スケールの分子動力学計算とシームレスに連携可能
デフォルト力場をAmber99に変更
マニュアルで調整した生体高分子残基の電荷を全モジュールプログラムおよびHomology Modeling Professional for HyperChemの全モジュールプログラム間で保持
最新ユーザーマニュアル追加
バージョン8.1.2から最新バージョン8.1.5で追加された機能
核酸をカウンターイオンで中和する機能
100,000原子数を超える拡張フォーマットHINファイル(HyperChemフォーマット)からPDBフォーマットにフォーマットレベルを下げることにより99,999原子以降のPDBカラムがずれる現象に対応
AutoDockまたはAutoDock Vina単独で実施したPDBQT形式のヒット化合物ファイルをDockViewerプログラムで認識できる機能
一部結合情報が誤って生成されるPDBQT形式の低分子化合物の構造を正しい結合情報でPDBQT形式に変換する機能
MolDimensionプログラムでこれまで不飽和結合扱いされていたアロマティックリングをアサインする機能
rDockやGNINAなどSDF形式でドッキング結果を出力するドッキングプログラムからのドッキング結果の解析、閲覧、絞り込み機能搭載(rDockやGNINAのドッキングモードを初期構造として引き続き、AutoDockまたはAutoDock Vinaでドッキングが実施できる)
今後追加予定の機能
Clusterバージョン(Virtual Screening System)のAutoDock Vinaインシリコスクリーニングインターフェイスにクラスター分散処理機能を追加予定(Docking Study with HyperChemオリジナルのCluster機能は開発当時から分散処理機能に対応済み)
タンパク-タンパクドッキングプログラムMEGADOCKインターフェイスの開発
rDockやGNINAのプリポスト機能の開発
リガンドベーススクリーニング機能の搭載(構造ベースファーマコフォア予測、リガンドベースファーマコフォア発生機能、コンフォメーション探索機能は開発当時から搭載済みのため、モジュールプログラムを追加するのみの段階)
マシンラーニングスクリーニング機能の搭載(タンパク記述子機能は開発当時から搭載済み、新たな高性能記述子の開発)
日本語マニュアル
パンフレット
Docking Study with HyperChem, Revision H1 (PDFファイル:1MB)
Docking Study with HyperChem, Revision G1 (PDFファイル:2MB)
旧パンフレットと併せてご利用ください
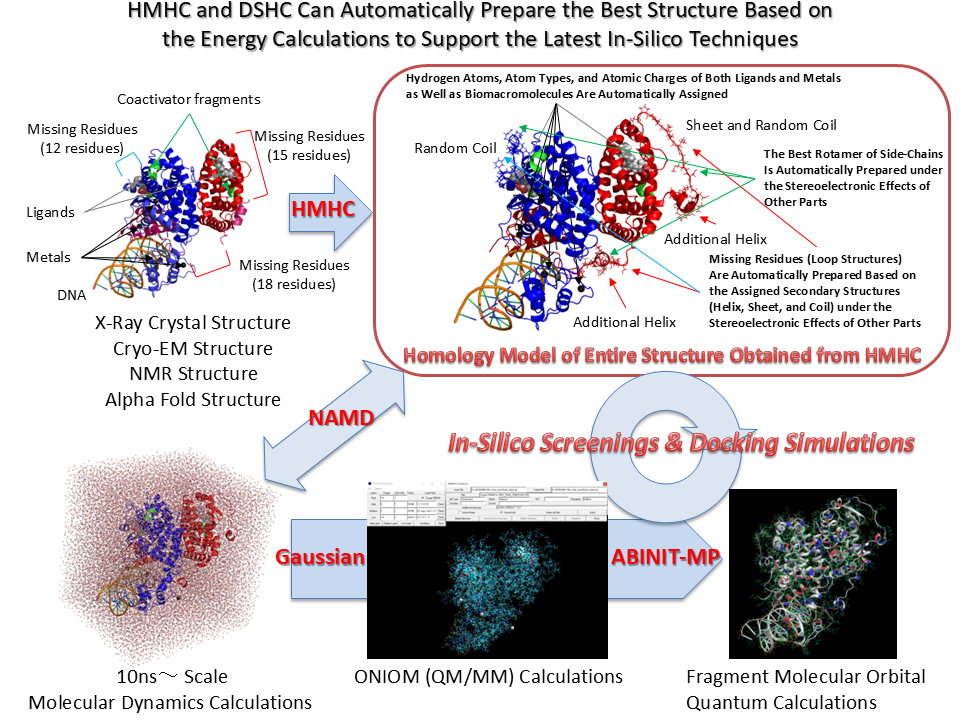
Homology Modeling Professional for HyperChem
with Gaussian Interface for HyperChem & ONIOM Interface for Receptor
Compatible to NAMD Molecular Dynamics and ABINIT-MP / GAMESS Fragment Molecular Orbital Quantum Mechanics Calculations for Entire Molecular System
旧リビジョンからの変更点
最新Gaussianプログラムに対応したONIOMインターフェイスを大幅機能強化
フラグメント分子軌道計算プログラムABINIT-MP(BioStation Viewer)およびGAMESS(Fu/Facio)用PDB形式への変換機能を搭載、全系量子力学計算プログラムとシームレスに連携可能
分子動力学計算プログラムNAMDのトラジェクトリからVMDで抽出したCHARMMベースのPDB形式を通常のPDB形式に変換する機能を搭載、数十ナノ秒スケールの分子動力学計算とシームレスに連携可能
デフォルト力場をAmber99に変更
マニュアルで調整した生体高分子残基の電荷を全モジュールプログラムおよびDocking Study with HyperChemの全モジュールプログラム間で保持
最新ユーザーマニュアル追加
バージョン8.1.2から最新バージョン8.1.5で追加された機能
核酸をカウンターイオンで中和する機能
100,000原子数を超える拡張フォーマットHINファイル(HyperChemフォーマット)からPDBフォーマットにフォーマットレベルを下げることにより99,999原子以降のPDBカラムがずれる現象に対応
低分子エディタでPDB中の低分子に水素原子付加した場合に自動的に水素原子名を生成する機能
残基名と残基番号が付与されているPDB中の低分子に水素付加した場合に付加した水素原子が別残基扱いされる機能を修正
今後追加予定の機能
NAMDインターフェイス(VMDに相当)の開発(バージョン8.1.0よりファイルフォーマットに対応済み)
AbInit-MPインターフェイス(BioStation Viewerに相当)の開発(バージョン8.1.0よりファイルフォーマットに対応済み)
日本語マニュアル
パンフレット
Homology Modeling Professional for HyperChem, Revision H1(PDFファイル:1MB)
Homology Modeling Professional for HyperChem, Revision G1(PDFファイル:4MB)
旧パンフレットと併せてご利用ください
構造ベース創薬(SBDD)システム資料
旧リビジョンに関しては以下を参照してください。
What's New 2015/01
What's New 2012/01
What's New 2010/02
What's New 2008/10
What's New 2008/05
What's New 2007/11
What's New 2007/02













