![]()
分子設計支援システム 生体高分子システムのためのGaussian ONIOM(QM/MM)計算用インターフェイス
ONIOM Interface for Receptor
- HyperChem GUIをコアとする、世界初完全自動インターラクティブマルチレイヤONIOM計算インターフェイス -
Compatible to Gaussian98, Gaussian03, Gaussian09, Gaussian16 & GaussView3, GaussView4, GaussView5, GaussView6
ワンクリックで生体高分子全系量子化学計算、タンパク質−リガンド複合体量子化学計算、核酸−リガンド複合体量子化学計算が実行できる世界初の生体高分子量子化学計算インターフェイス
最先端のインシリコ創薬において、生体高分子システムなどの巨大分子システムに対して全系量子力学計算を実施する場合、分子力学計算による構造最適化計算や分子動力学計算で用意した初期構造では一点計算さえ収束しないか、異常なエネルギー値を与えます。フラグメント分子軌道法などで全系量子力学計算を実施する場合は、ONIOM法で予め初期構造を更に構造最適化して置く必要があります。ONIOM Interface for ReceptorはHomology Modeling Professional for HyperChemやDocking Study with HyperChemで準備した高精度初期構造(複合体構造)を量子力学計算用に更に精密化して全系量子力学計算を成功させる最善のソリューションを提供します。
Motonori Tsuji*, Koichi Shudo, Hiroyuki Kagechika. FEBS Open Bio., 7, 391-396, 2017, DOI: 10.1002/2211-5463.12188.
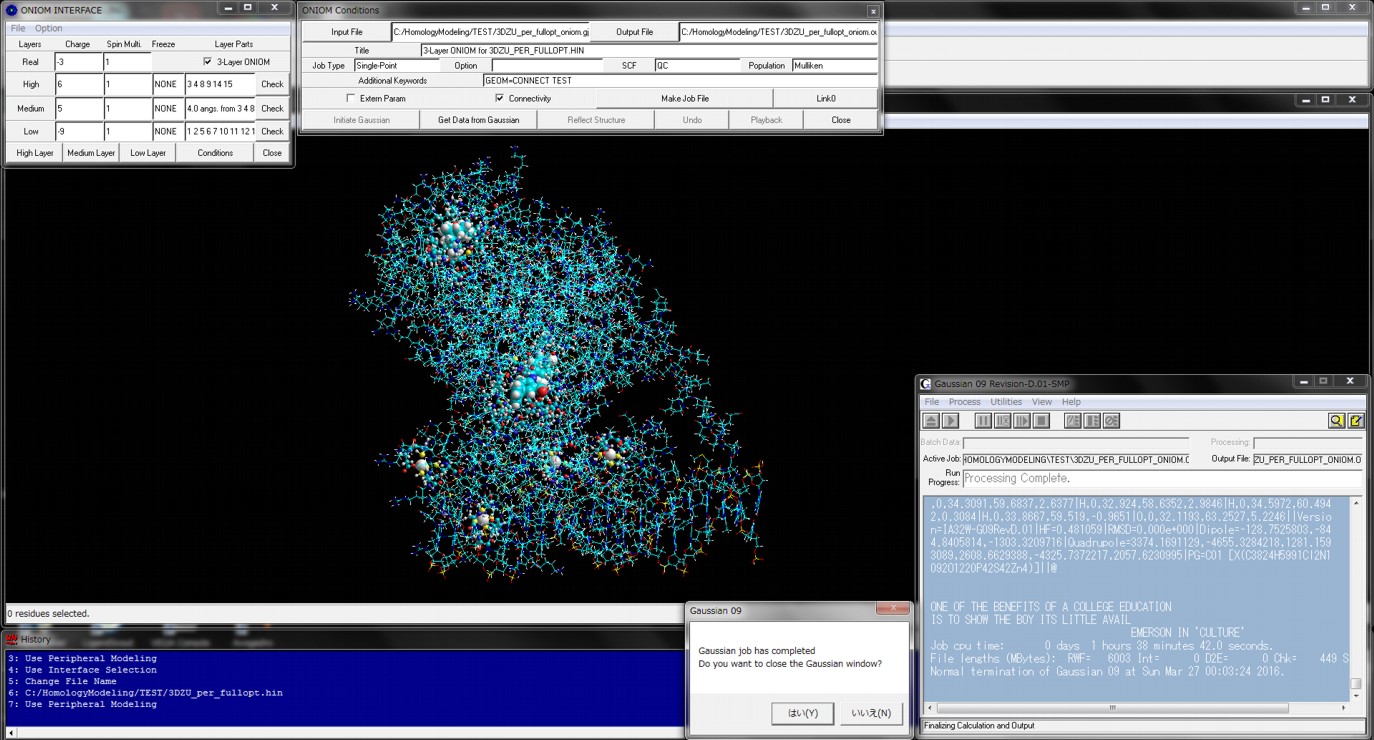
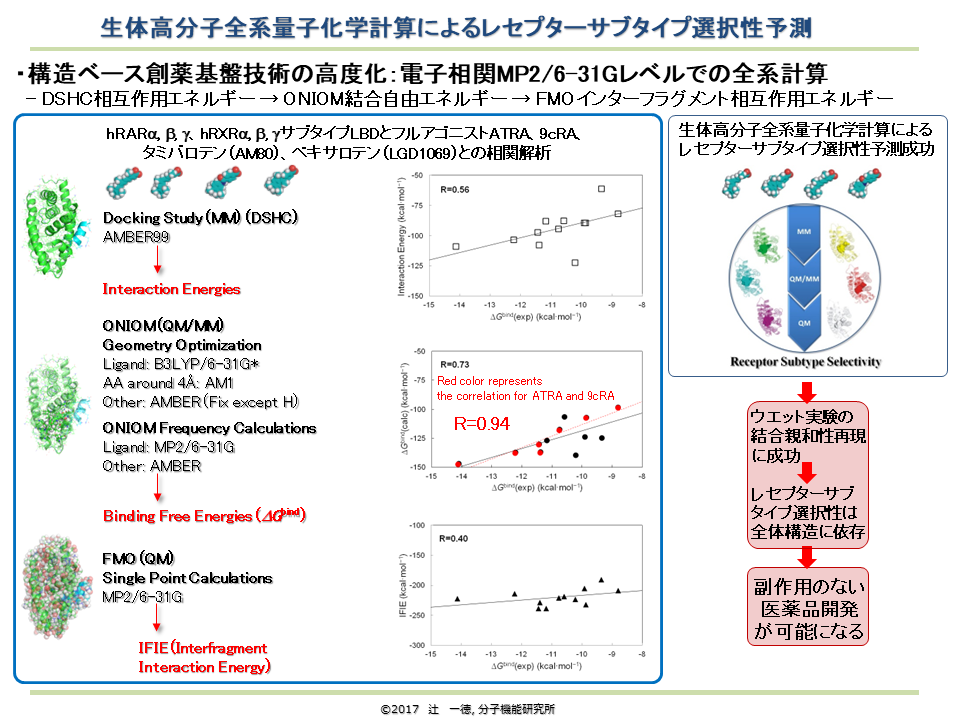
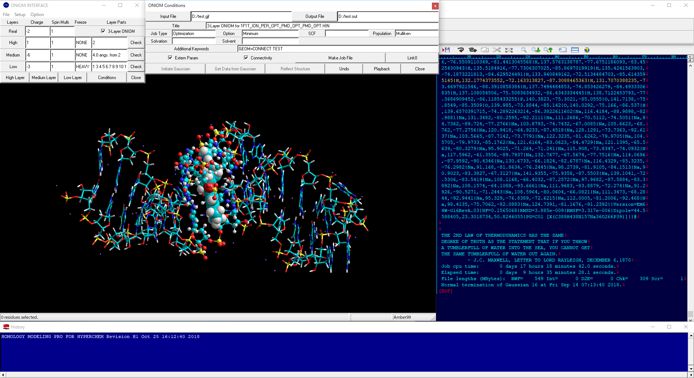
上図は核内受容体DNA結合領域−リガンド結合領域のヘテロダイマーとコファクターフラグメントおよびDNA上のホルモン応答配列とのジンクフィンガーモチーフ全系に対する3レイヤONIOM計算(RHF/6-31G:RAM1:AMBER)の結果です。Highレイヤには2種類のリガンドと4個のZn原子を設定し、MediumレイヤにはHighレイヤから4Å内の残基およびヌクレオチドと水分子とし、残りの構造をLowレイヤに設定しています。計算に必要なパラメータは全て自動で設定されるため、ONIOMインターフェイス起動と同時にGaussianを実行できます。本計算はWindows版Gaussianで行っていますが、UNIX64ビットマルチプロセッサ版Gaussian用ジョブファイルの準備に威力を発揮します。
世界が認める最強の分子モデリング環境
HyperChem front-ended
世界が認める最強の計算化学環境
Gaussian back-ended
本製品はGaussianオフィシャルサイトにトップレベルでリンクされています。
特徴
タンパク分子システムなどの巨大分子システムに対する代表的な量子化学計算手法として、現在、Gaussianが搭載するONIOM法、MOPAC200Xが搭載する半経験分子軌道法によるMOZYME法およびAb-InitioフラグメントMO法が挙げられます。本プログラムはHyperChemユーザーインターフェイスを利用してGaussianに搭載されるONIOM計算を極めて容易に実行可能にします。また、HyperChemユーザーインターフェイスを利用してMOPAC2000に搭載されるMOZYME計算を実行できるMopac Interface for HyperChemが利用可能です。更に、最新版ではフラグメント分子軌道法(FMO)計算プログラムABINIT-MP(BioStation Viewer)やGamess(Fu/Facio)用PDB互換機能を搭載しています。したがって、本プログラムの登場により、巨大分子システムに対する代表的量子化学計算手法の全てがHyperChemユーザーインターフェイス上で極めて容易に実行可能となります。
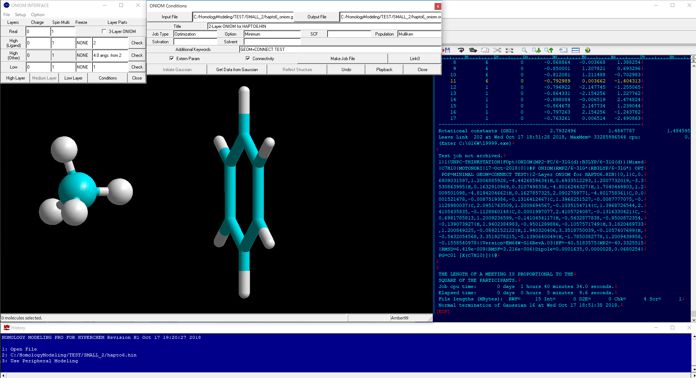
ONIOM Interface for ReceptorはHyperChemの分子モデリング機能で用意した分子システムの2-レイヤ(QM:QMおよびQM:MM)および3-レイヤONIOM(QM:QM:QMおよびQM:QM:MM)計算のためのGaussianジョブファイルを起動後、僅か数秒で、パラメータを一切編集することなく、自動的に生成してGaussianを起動し、その計算結果を再びHyperChemに取り込むことができます。HyperChemに取り込める情報としては、各種電荷および計算構造です。さらに、構造最適化中の途中構造をリアルタイムで更新できます。ONIOM Interface for Receptorはレセプターとリガンドなどの生体高分子(タンパクや核酸)と低分子からなる分子システムに特化したONIOMインターフェイスです。このため、Gaussianにおける不足力場パラメータ、リンク原子情報および電荷情報を自動的に生成できます。また、HyperChemに計算結果をその場でフィードバックさせる必要がない場合には、UNIX版Gaussian用ONIOMインターフェイスとしても極めて有用です。
本プログラムは分子モデリング、分子エディタモジュールプログラムである周辺モデリングプロフェッショナルモジュールプログラムにGaussian Interface for HyperChemとともに一体化されており、それにより低分子化合物との複合体構造や金属原子を含む巨大分子システム全体を量子化学的に処理して分子設計、相互作用解析、遷移状態解析、機能解析できるようになります。
具体的な使用法は実施例を参照してください。
GaussViewにONIOM Interface for Receptorで自動作成したジョブファイルや結果ファイルを読み込ませることで、連携し、より高度な計算が簡便に行えます。
*ONIOM Interface for ReceptorのLink0オプションにあるCheckpointファイル入力ボックスにファイル名を指定すれば、GaussianのCheckpointファイルが生成されますので、CheckpointファイルあるいはFormcheckファイルを必要とする他のGaussianビューアで分子軌道や静電ポテンシャル等の計算結果を確認することができます。
**(QM:MM)および(QM:QM:MM)の場合、Lowレイヤで設定できる分子力場計算(MM)はAmberのみです。GaussViewにジョブファイルを読み込ませることでGaussianがサポートするその他の力場に容易に変換できます。
以下はONIOM Interface for Receptorを介してGaussianの2-レイヤONIOM(HF/6-31G*:Amber)で構造最適化(赤色)し、その構造をオリジナルの構造(青色)とタンパク重ね合わせプログラムでRMSフィットした結果です。このサイズでもONIOM構造最適化計算は3時間程度(32ビットシングル版Gaussian03W)でした。
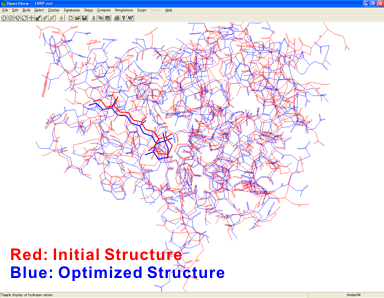
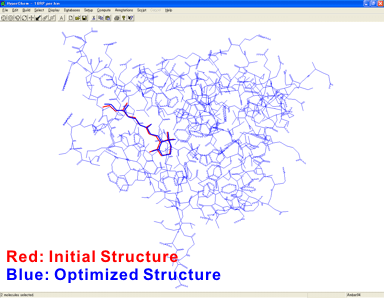
以下は周辺モデリングプロフェッショナルモジュールプログラムで調整した初期構造(赤色)をもとにGaussianの2-レイヤONIOM(HF/6-31G*:Amber)で構造最適化(青色)し、その構造をHyperChemにフィードバックしてタンパク重ね合わせプログラムでRMSフィットした結果です(右図はLowレイヤの重原子座標を固定した部分構造最適化の結果です)。
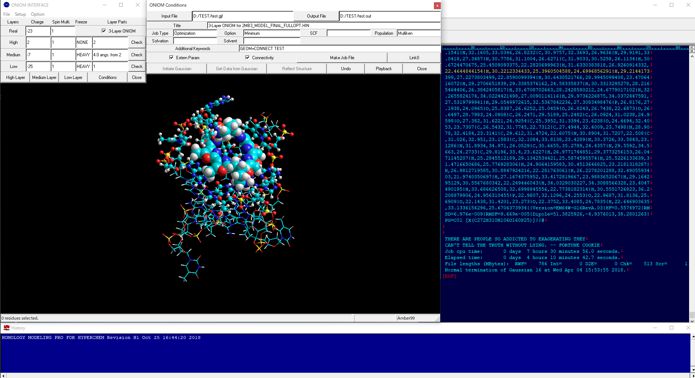
複雑な生体高分子システムのONIOM計算(量子化学計算)もワンクリックで実施できます。生体高分子−リガンド複合体構造がPDBに登録されているケースはもちろん、Docking Study with HyperChemで高精度ドッキングシミュレーションを実施して得た安定複合体構造の精密化などでも威力を発揮します。ONIOM(量子化学)構造最適化計算の後にはフラグメント分子軌道法のためのファイル互換機能も搭載されており、全系量子化学計算をシームレスに実施することが出来ます。MP2などの摂動法をQMに適用することで電子相関を考慮でき、振動解析を実施すればMP2電子相関レベルでの第一原理量子化学計算や密度汎関数法による非経験的結合自由エネルギーを算出することもできます。
下図は抗原抗体反応の2-layer ONIOM計算の結果です。抗原をハイレイヤ(HF/6-31G)に設定して完全構造最適化を実施し、2分子の抗体と176分子の結晶水をロウレイヤ(AMBER)に設定して水素原子のみ構造最適化した結果です。
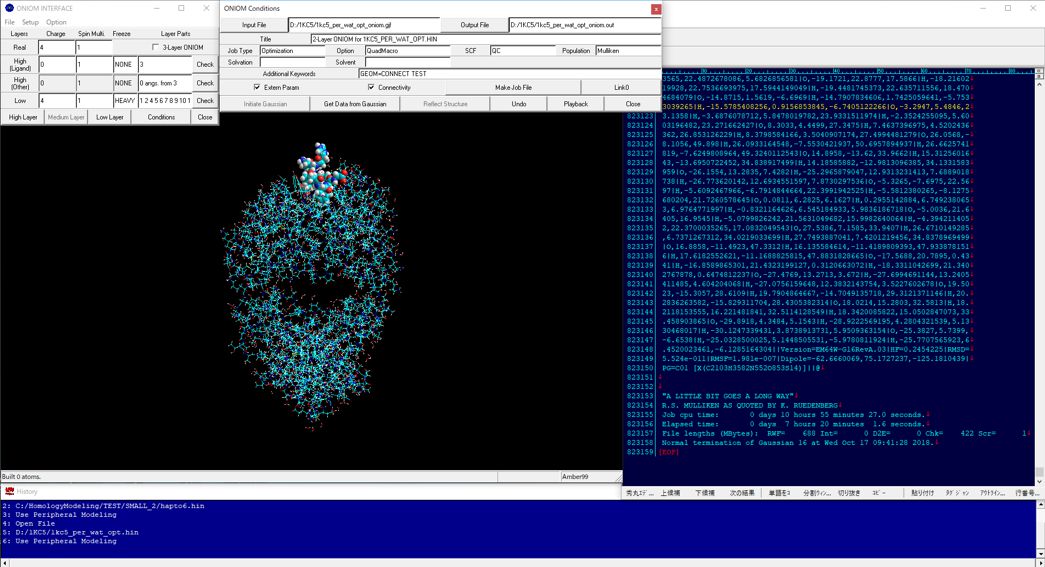
下図はハプト6型のCH-パイ相互作用の2-layer ONIOM計算です。ハイレイヤ(MP2/6-31G*)にメタンを設定し、ロウレイヤ(B3LYP/6-31G*)にベンゼンを設定して全体を完全構造最適化した結果です。
下図はDNAと中分子の3-layer ONIOM計算の結果です。ハイレイヤ(HF/6-31G*)に中分子を設定して完全構造最適化し、ミディアムレイヤ(AM1)に中分子から4Å内のヌクレオチドを設定、残りのDNAをロウレイヤ(AMBER)に設定して水素原子のみ構造最適化した結果です。
下図はRNAと低分子の3-layer ONIOM計算の結果です。ハイレイヤ(B3LYP/6-31G*)に低分子を設定、ミディアムレイヤ(AM1)に低分子から4Å内のヌクレオチドを設定して完全構造最適化し、残りのRNAとカウンターイオンをロウレイヤ(AMBER)に設定して水素原子のみ構造最適化した結果です。
製品紹介論文
Motonori Tsuji. Molecular Science, 1, NP004, 2007.
成果学会報告
成果論文
Microbiol. Spectrum. 12, 2024.
J. Autoimmun. 139, 103085, 2023.
Motonori Tsuji*. Int. J. Mol. Sci. 23, 11009, 2022.
PLOS ONE 16, e0257705, 2021.
Chem. Pharm. Bull. 68, 1193-1200, 2020.
J. Virol. 2020.
Motonori Tsuji*. FEBS Open Bio., 10, 995-1004, 2020.
J. Pharmacol. Exp. Ther. 372, 277-284, 2020.
Motonori Tsuji.* J. Comput. Aided Mol. Des., 31, 577-585, 2017, DOI: 10.1007/s10822-017-0025-6.
Motonori Tsuji*, Koichi Shudo, Hiroyuki Kagechika. FEBS Open Bio., 7, 391-396, 2017, DOI: 10.1002/2211-5463.12188.
Motonori Tsuji.* J. Mol. Graph. Model., 62, 262-275, 2015.
Motonori Tsuji*, Koichi Shudo, Hiroyuki Kagechika. J. Comput. Aided Mol. Des., 29, 975-988, 2015.
Motonori Tsuji.* J. Struct. Biol, 185, 355-365, 2014.
Chem-Bio Informatics Journal, 12, 1-13, 2012.
Plant Cell Physiol. 2012 Jul; 53(9): 1638-1647.
Biochemistry. 2010 Dec; 49(50): 10647-10655.
Biochemical and Biophysical Research Communications. 2010 Apr 30; 395(2): 173-177.
Science. 2008 Feb 1; 319(5863): 624-7.
備考
ONIOM Interface for ReceptorはHomology Modeling Professional for HyperChemの分子エディタモジュールプログラムである周辺モデリングプロフェッショナルモジュールプログラムに付属しています。
ONIOM Interface for Receptorは2分子以上が作業領域に存在しないと有効になりません。
最新、高性能・高機能生体高分子システムモデリング、機能解析、シミュレーションパッケージ
Homology Modeling Professional for HyperChem
with Gaussian Interface for HyperChem & ONIOM Interface for Receptor
Compatible to NAMD Molecular Dynamics and ABINIT-MP / GAMESS Fragment Molecular Orbital Quantum Mechanics Calculations for Entire Molecular System
*HyperChemはHypercube, Incの登録商標です。
**GaussianはGaussian, Inc.の登録商標です。
本製品の開発にあたり、Hypercube, Inc.およびGaussian, Inc.の承認済みです。













