![]()
Homology Modeling Professional for HyperChem
![]()
Restraints(束縛)
特徴
低分子に対しては、分子動力学計算は安定コンフォメーションの探索あるいは複数の初期構造の生成を目的として使用し、ほとんどの場合に束縛条件の必要性はありません。しかしながら、生体高分子などの巨大分子に対する分子動力学シミュレーションあるいはアニーリングには束縛条件設定が大前提の必須となります。そこで問題となるのは、その束縛条件の膨大な数と正確な設定です。通常はこれを手動で膨大な時間をかけて入力ファイルとして作成しますが、本モジュールプログラムを利用することでこの問題をいっきに解決できます。
束縛モジュールプログラムはタンパクにおける二面角(φ、ψおよびアミド)とタンパク質分子、核酸分子、水分子および、その他のヘテロ分子の全原子座標(主鎖原子や側鎖原子などのグループも可能)に構造最適化あるいは分子動力学計算のための束縛条件をいっきに、そして個別の設定で準備できるこれまでにない画期的なユーザーインターフェイスです。通常は、モデル作成最終段階での分子動力学アニーリングに必要な束縛条件設定のために使用しますが、その有用性は既存の使用法に限定されず、例えば、アポタンパクからホロタンパクへの反応座標を追跡するなど、生体高分子の動力学的挙動を完全にGUIベースで制御できます。HyperChemに搭載される全ての分子動力学計算、Molecular Dynamics、Langevin Dynamics、Monte Carloとともに、構造最適化もサポートされています。また、HyperChemに搭載される全ての力場、Ambers、Amber2、Amber3、Amber94、Ambre96、Amber99、MM+、OPLS、Bio+83、Bio+85、CHARMM19、CHARMM22、CHARMM27、をサポートしています。
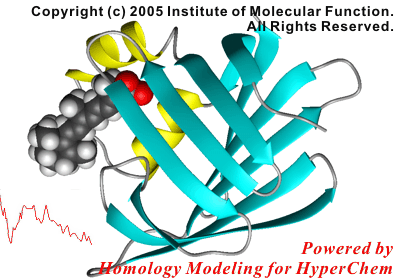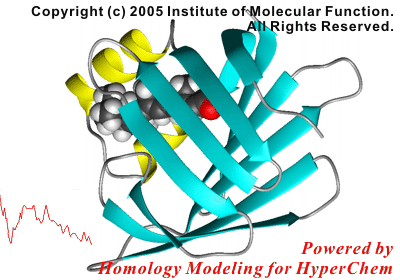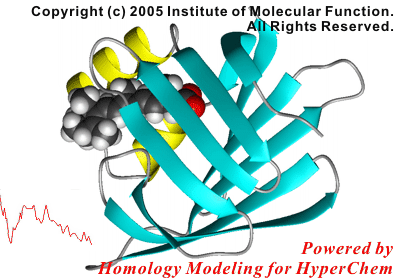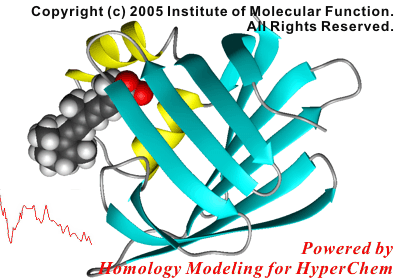
以下は本機能を利用したリガンドエントリーに関する反応座標解析研究の結果です。