![]()
人レチノイドXレセプターアゴニスト設計
論理的構造ベース薬物設計:RXRアゴニスト設計、リード最適化
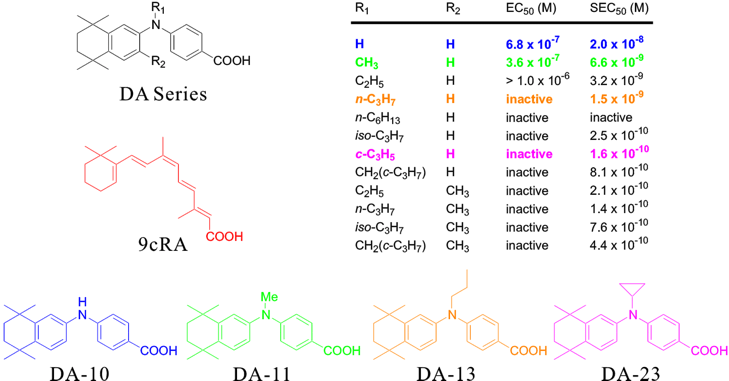
以下にジフェニルアミン誘導体、DAシリーズ、のレチノイン酸レセプター、RAR(EC50)およびレチノイドXレセプター、RXR(SEC50)に対する構造活性相関を示す。
窒素原子上の置換基、R1、がH(DA10)あるいはメチル基(DA11)では若干低いRXRアゴニスト活性とともに弱いRARアゴニスト活性も見られる。これに対して、R1が嵩高くなるにつれて非常に強いRXR選択的アゴニストとなることが分かる。しかしながら、R1がC3単位以上になると逆に両レセプターへの親和性が失われることが分かる。*
*Biol. Pharm. Bull., 1998, 21, 544-546.
論理的構造ベース薬物設計 − リード最適化手順
準備:レセプターの構造はHomology Modeling Professional for HyperChemリビジョンB1を用いて調整、準備した。各化合物の安定コンフォメーションはDocking Study with HyperChemに付属する自動分子2D-3D変換プログラム、Mol Dimensionにより準備した。レセプターとのフレキシブルドッキングシミュレーションはプロフェッショナル版Docking Study with HyperChemリビジョンA3を用いてAmber94力場、All Atom条件下に実施した。
以下に代表的な化合物に対するAF2アゴニストコンフォメーションを採用するRXRαリガンド結合領域立体構造(水分子およびコアクチベータフラグメントを含む)とのタンパク・リガンドフレキシブルドッキングシミュレーション(レセプターおよび化合物ともに動的に取り扱う)の結果を示す。
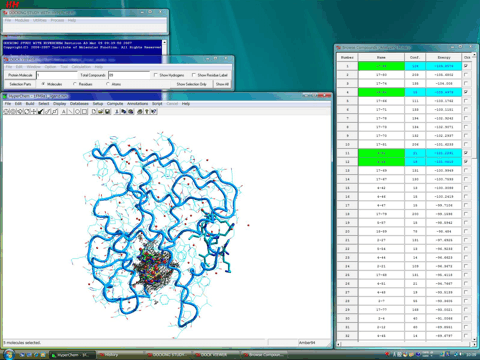
下図は、相互作用エネルギー(表中相互作用エネルギーはソートされている)の観点から、フレキシブルドッキングシミュレーションで得た各化合物の最も安定な複合体結合様式を示しており、複合体としてX線結晶構造解析された天然リガンドの9-シスレチノイン酸(赤;9cRA)の構造を同時に示している。またフレキシブルに取り扱ったタンパク側のアミノ酸側鎖をチューブで示しており、非常に良好なインデュースドフィット効果(誘導適合効果)が認められる。さらに、相互作用エネルギーは実験で得られた活性データと極めて良好な相関(構造活性相関)を示した。
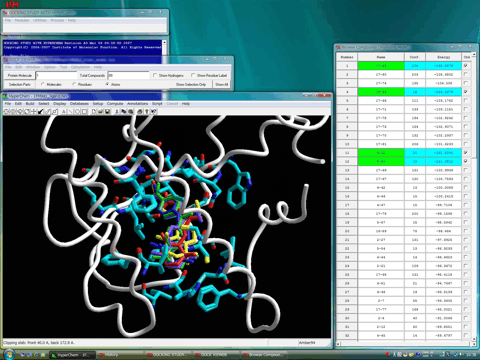
また、下図はリガンド結合部位のファンデルワールス表面を同時に示している。
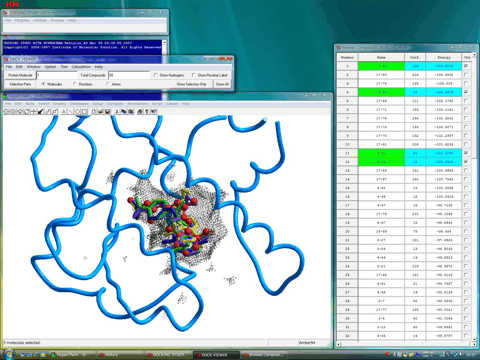
ドッキングシミュレーションの結果、今回検証したDAシリーズはいずれも安定複合体では9cRAとほぼ同様の結合様式を採用していた。
また、構造活性相関に重要な窒素上置換基は9cRAの9位のメチル基に対応することがわかる。
窒素上に置換基を持たないDA10(左上;青)の複合体はキャビティーに余裕が残されており、ジフェニルアミン骨格全体がキャビティー面に向かってシフトしていた。したがって、DA10ではキャビティー内部の疎水性残基とのトータルなファンデルワールス接触が少なく、ファンデルワールス力による複合体安定化の効果が他の化合物よりも小さいと考えられ、比較的低いアゴニスト活性を説明できる。
これに対して、n-プロピル基を持つDA13(左下;黄)はn-プロピル基とキャビティー内部の疎水性残基との接触が多く、ファンデルワールス力による複合体安定化の寄与が大きいが、若干の立体反発が見受けられた。つまり、化合物立体構造が幾分かひずんでおり、フェニル基同士がスタッキングコンフォメーションに近いコンフォメーションとなっていた(他はT-シェイプコンフォメーションに近い)。これにより、n-プロピル基が窒素上置換基の限界の大きさであるという実験結果を説明できる。
窒素上置換基がメチル基であるDA11(右上;緑)およびシクロプロピル基であるDA23(右下;ピンク)ではリジッドに安定な複合体を形成していた。
ただし、DA11ではDA13と比較してキャビティー上部にC2-C3単位分の余裕があり、この分のファンデルワールス力による安定化の減少がDA11の比較的低いアゴニスト活性に関係があると推測される。
DA23の最も強いアゴニスト活性は、嵩高い疎水性置換基(シクロプロパン)がトータルなファンデルワールス接触を増大させ、かつ化合物の立体構造にひずみをもたらすほどのレセプターとの接触がないことでファンデルワールス力が有効に複合体の安定化に寄与したためと予想される。
この観点からすると、シクロプロピル基をもつDA23でもキャビティー上部に若干の余裕があり(DA13と比較して)、シクロプロピル基1位へのC1-C2単位程度の疎水基の導入により、さらに活性が向上するものと推測される。
2005年11月6日













