![]()
Homology Modeling for HyperChem
詳細な手順、設定値、および分子動力学アニーリング等を含むHyperChemオプションの設定手順、設定値等は付属のマニュアルのチュートリアルに詳細に示されています。
ホモロジーモデリングチュートリアル(グロビュラータンパク質、核内受容体:人ミネラルコルチコイドレセプターリガンド結合領域)
使用プログラム:Homology Modeling for HyperChem Revision B1
2007年
1.コントロールセンターを起動し、X線結晶構造解析された人グルココルチコイドレセプターリガンド結合領域の構造を鋳型としてロードする。
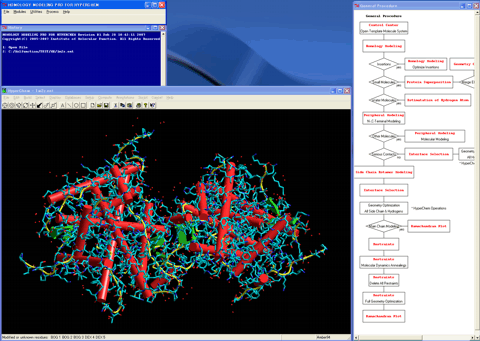
2.ホモロジーモデリングモジュールプログラムを呼び出し、標的配列である人ミネラルコルチコイドレセプターアミノ酸配列をロードする(デフォルトではBlosum62によるペアワイズアラインメントが実行され、鋳型タンパクに対して二次構造部分が判定される)。
3.ホモダイマーを形成している鋳型タンパク質分子システムの各タンパク質分子の熱振動および標的アミノ酸配列とのアミノ酸一致度から、適切なタンパク質分子を鋳型に決定する。
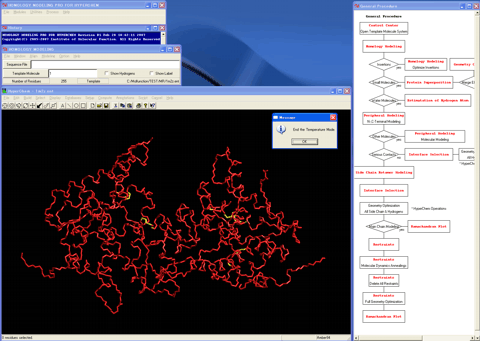
4.必要に応じてアライン最適化を実施する。
5.必要に応じて標的アミノ酸配列におけるヒスチジン残基のプロトン化状態をアサインする。
6.挿入配列モデリングのための計算条件を設定し、標的アミノ酸配列の挿入配列部分が採りうる二次構造またはランダムコイルをアサインする。
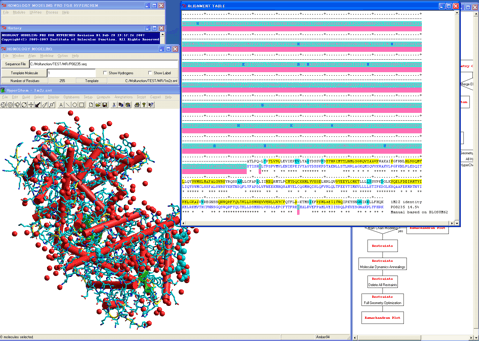
7.初期3Dモデルを作成する。
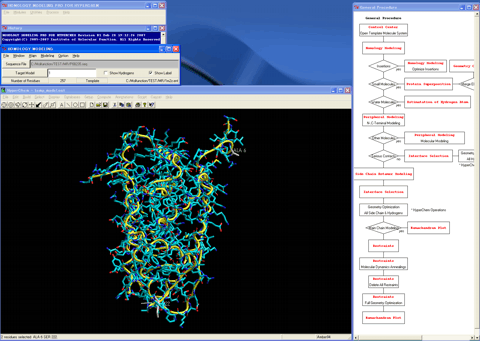
8.タンパク重ね合わせモジュールプログラムを呼び出し、鋳型タンパク質分子システム(青色)を初期3Dモデル(赤色)にマージして重ね合わせる。
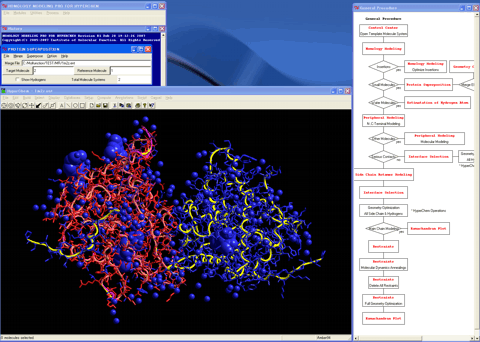
9.インターフェイス選択モジュールプログラムを呼び出し、マージした鋳型タンパク質分子システムからモデルに接触する水分子、リガンドやコファクターなどの重要な成分を抽出して、モデルに取り込む。
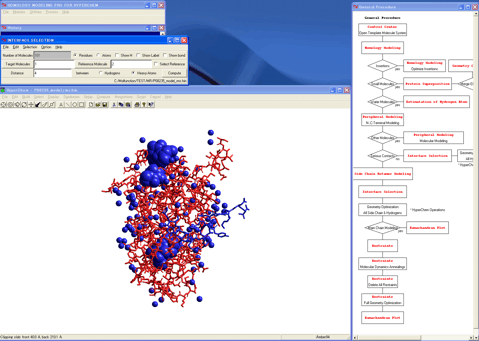
10.水素原子初期座標評価モジュールプログラムを呼び出し、取り込んだ水分子における水素原子の最善の初期座標を生成する。
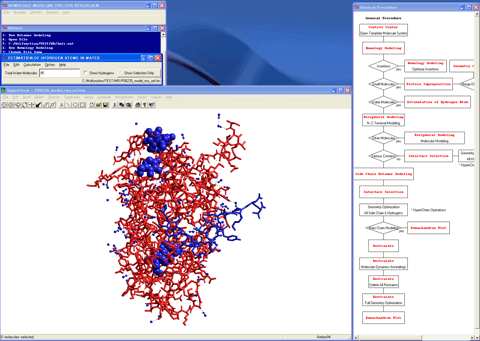
11.周辺モデリングモジュールプログラムを呼び出し、以後の作業用力場条件をセットする。
12.必要に応じて末端残基モデリングモードにて各タンパク質分子の末端残基をツビッターイオンに変換しておく。
13.低分子モデリングモードにする。
14.鋳型から抽出したリガンドに結合情報および水素原子を付加して原子タイプを割り振る。
15.Gaussianインターフェイスを呼び出し、作業用力場とバランスの取れた計算理論の一点計算を実施し、結果をフィードバックしてリガンドに電荷を割り振る。
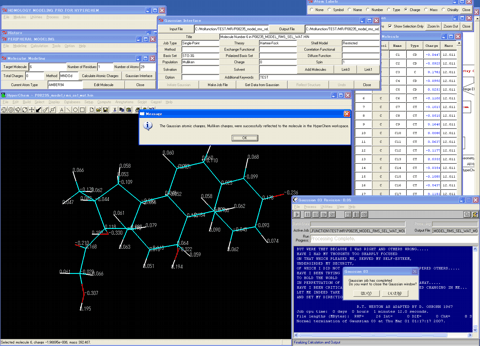
16.その他の低分子を含んでいる場合には、これらについても結合情報および水素原子を付加して原子タイプと電荷を割り振っておく。
17.側鎖ロータマーモデリングモジュールプログラムを呼び出し、最終アラインメントをロードして反映させる。
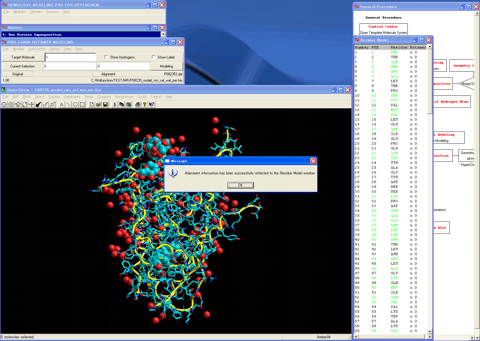
18.作業用力場条件および自動ロータマー探索条件をセットし、バッチ計算にて鋳型と異なる側鎖の最良のロータマーを決定する。
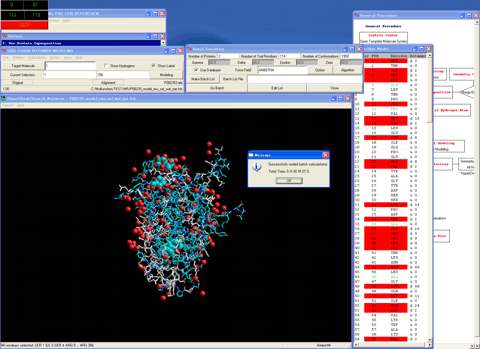
19.インターフェイスモジュールプログラムを呼び出し、モデルの側鎖および全分子の水素原子を選択する。
20.作業用力場条件下に選択原子のみを対象に構造最適化する。
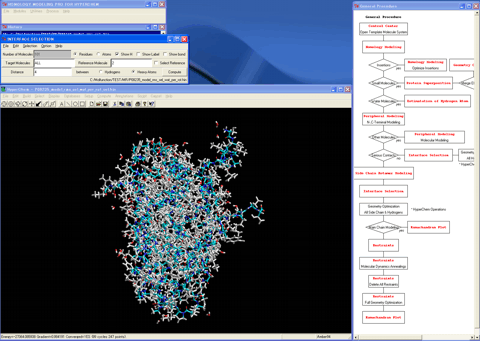
21.束縛モジュールプログラムを呼び出し、全重原子を対象に束縛条件をセットする。
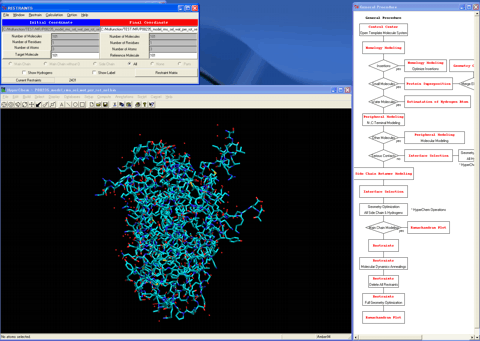
22.作業用力場条件をセットし、低温分子動力学アニーリングを実施する。
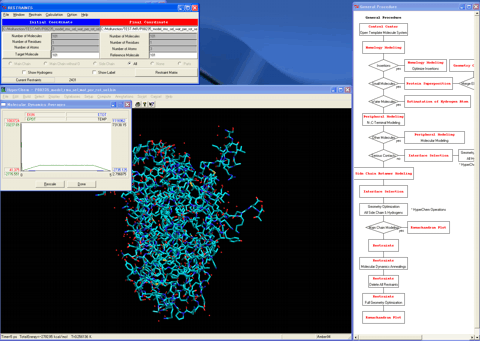
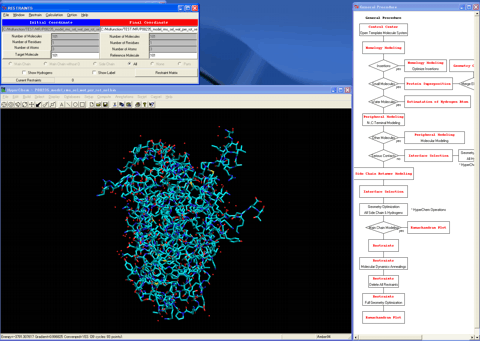
23.全束縛条件を削除し、構造最適化を実施する。
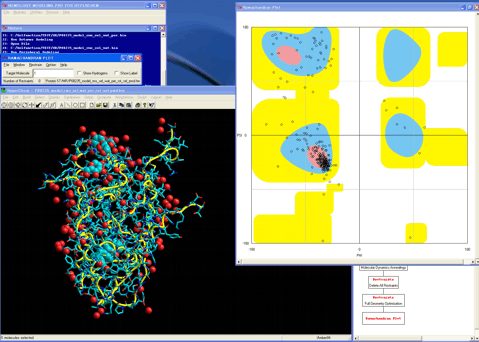
24.ラマチャンドランプロットモジュールプログラムを呼び出し、モデルの精度を確認する。
ホモロジーモデリング終了。
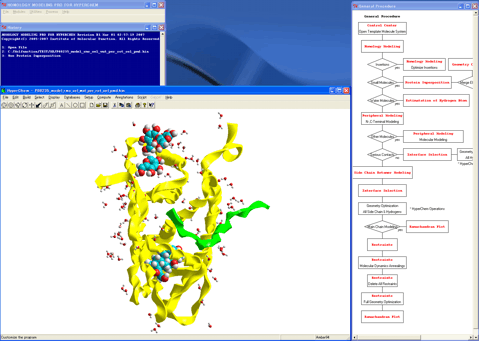
通常数日から数週間かかる作業が約2時間で終了します。しかも、すべてエネルギー計算に基づいているため論理性、再現性、網羅性が保証されます。その上で、各モデリング過程で研究者のロジックをモデルに反映させることができます。













